Unterschied zwischen der gesamten Genomsequenzierung und der Schrotflintensequenzierung
- 3828
- 1169
- Prof. Dr. Dylan Hentschel
Was ist die Sequenzierung des gesamten Genoms und die Schrotflinte -Sequenzierung??
Dies sind Methoden zur Beurteilung und DNA -Sequenzen eines Organismus -Genoms zu einem einzigen Zeitpunkt.
Ähnlichkeit
Ähnlichkeiten zwischen der gesamten Genomsequenzierung und der Schrotflintensequenzierung umfassen;
- Beide sind an der Sequenzierung von Desoxyribonukleinsäure -Fragmenten beteiligt
- Beide Techniken beinhalten eine genomische Fragmentierung
- Beide Techniken verwenden die Digitalisierung und basieren Computer
- Beide Techniken spielen eine entscheidende Rolle in Forschungsstudien und in der molekularen Diagnostik

Ganzes Genomsequenzierung
Die Sequenzierung des Gesamtgenoms alias (WGS) ist eine detaillierte Technik zur Untersuchung der gesamten genomischen Desoxyribonukleinsäuresequenz einer Zelle zu einem einzigen Zeitpunkt.
Die Sequenzierung des gesamten Genoms wurde nach der Veröffentlichung des Human Genom-Projekts verfügbar, das die Referenz für menschliche Genomsequenzen lieferte.
Schrotflintensequenzierung
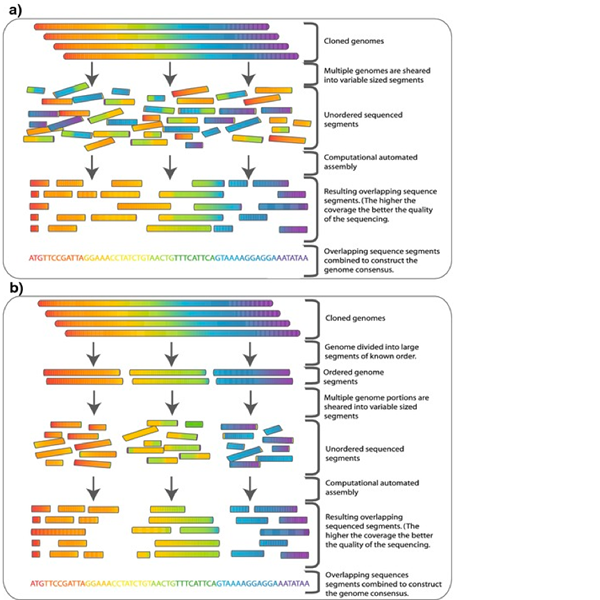
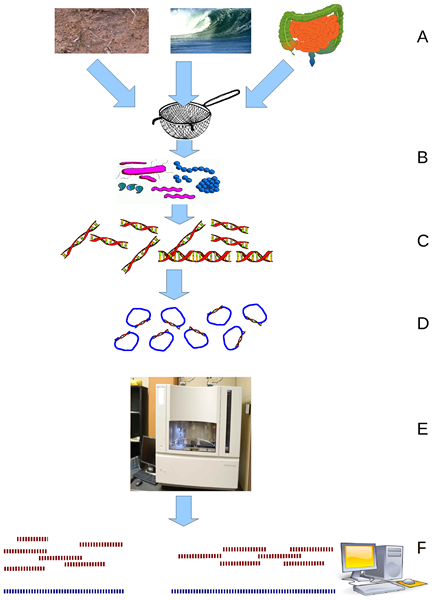
Die Schrotflintensequenzierung beinhaltet die ungeplante Aufteilung von Desoxyribonukleinsäure -Aufteilung von Desoxyribonukleinsäuresequenzen in viele kleine Fragmente und montiert sie dann erneut (die Sequenz), indem sie die Regionen der Überlappung herausgefunden haben.
Unterschied zwischen der gesamten Genomsequenzierung und der Schrotflinte
Beschreibung
Ganzes Genomsequenzierung
Ganzes Genomsequenzierung (WGS) ist ein Ein-Schritt-Anordnungsprozess. In dieser Technik findet zuerst das gesamte Genom (haploides Chromosomen in einem Mikroorganismen) Scheren statt. Danach wird jeder dieser winzigen spröden Scherben zufällig einem Anordnungsprozess unterzogen. Sobald diese Anordnung oder Sequenzierung abgeschlossen ist, erfolgt die Analyse. Zum Zeitpunkt der Beurteilung der Anordnung/Sequenzen wird ein überlappender Sequenzierungsprozess beseitigt, und die Bewertung ist keine geordnete Technik. Daher ist die Montage von Sequenzen ein weniger erfolgreicher und wirksamer Prozess. Der Prozess der Sequenzierung des gesamten Genoms ist jedoch Swifter, und die Beurteilung des gesamten Genoms (haploiden Chromosomen in einem Mikroorganismen) kann in einer einzigen Instanz stattfinden.
Schrotflintensequenzierung
Die Schrotflintensequenzierung ist eine Labormethode zur Bewertung der Desoxyribonukleinsäuresequenz eines genetischen Materials eines Organismus (Genom). Die Technik beinhaltet die Aufschlüsselung des genetischen Materials des Organismus in winzige Desoxyribonukleinsäure -Fragmente, die die individuelle Sequenzierung darstellen.
Verwendet
Ganzes Genomsequenzierung
- Neugeborene und pädiatrische Erkrankungen - zieht strukturelle Varianten ab und scannt exonische Bereiche, die nicht angemessen erfasst werden.
- Arzneimittelversuche und Pharmakogenomik - WGS bietet einen vorteilhaften Front -End -Mechanismus für klinische Studien, die bei der Klassifizierung von Personen (Patienten) aufgrund ihrer Reaktion auf das untersuchte Medikamente oder Medikamente beitragen.
- Regulatorische Variation und EQTLs
- Seltene Gerüchttypen - WGS ist die richtige Methode, um seltene Tumoren zu untersuchen. Die Technik hilft bei der Untersuchung und Aufnahme des gesamten Mutationsbereichs, von einzelnen Basenänderungen (eine einzelne Basis in der Desoxyribonukleinsäure unterscheidet.
- Clan -Genomik - Stammbäume für Familienkrankheiten - TAPS -Multiplex -Stammbäume aus Familien, die von genetischen Problemen betroffen sind.
- Große Kohorten mit umfangreichem Phänotypisierung
Schrotflintensequenzierung
- Untersucht die Desoxyribonukleinsäuresequenz des Genoms eines Organismus.
- Effiziente Methode, um Ribonukleinsäure oder Proteine indirekt zu sequenzieren (über ihre offenen Leserahmen)
- Die am meisten bevorzugte Methode für alle anderen Arten von Haploid -Satz von Chromosomen in einer Mikroorganismus -Sequenzierung. Der gesamte Haploid -Satz von Chromosomen vieler Organismen: Kuh, Hühnchen, Ratte, Pflanze Arabidopsis thaliana, Kugelfische und viele mikroskopische Organismen wurden nach dieser Technik sequenziert.
- Nahe und fast endgültige De-novo-Assemblys von mikrobiellen Genomen in sehr weniger Tagen
- Die Kraft zur Sequenz großer, komplexer Genome
- GS FLX+ System -Leselänge bis zu 1 kb
- Führen Sie Hybridbaugruppen durch
Schritte
Ganzes Genomsequenzierung
- Generierung der Sequenzierung liest direkt aus einem ganzen Genom (haploiden Chromosomen in einem Mikroorganismus) Repository
- Anwendung von Rechenmethoden, um sich in einzelnen Schritt wieder zusammenzustellen
- Wird für die Fruchtfliege (Drosophila melanogaster) und durch Celera -Genomik (entwickelt 1998) für menschliches Genom (haploides Chromosomensatz in einem Mikroorganisum) verwendet
Schrotflintensequenzierung
- Schritte in der Schrotflintensequenzierung umfassen;
- Bibliotheksvorbereitung
- Sequenzierung der cfDNA -Fragmente
- Fragmentzählung
- Sequenzausrichtung
- Statistische Analyse und Berichterstattung.
Zusammenfassung
Die Unterschiede zwischen der gesamten Genomsequenzierung und der Schrotflintensequenzierung wurden wie unten zusammengefasst:
Ganzes Genomsequenzierung gegen Schrotflintensequenzierung

- « Unterschied zwischen Sinovac -Impfstoff und Pfizer
- Unterschied zwischen orthopädischer Chirurgie und Neurochirurgie »

